Équipe
Épigénétique & Chromatine
Dpt: Signalisation et Chromatine
Nos activités de recherche
En contrôlant l’expression des gènes, l’organisation de la chromatine définit les identités cellulaires et constitue une barrière à la reprogrammation. Or, suite à des blessures, chaque espèce animale possède un potentiel spécifique pour réaliser de la régénération à grande échelle, impliquant souvent une régression de cellules somatiques à un stade indifférencié, puis leur prolifération et re-différentiation. Puissants pour maintenir l’homéostasie tissulaire, ces mécanismes sont aussi dangereux car ils offrent une fenêtre d’opportunité pour que certaines cellules échappent à leur identité programmée. Notamment, les cellules cancéreuses acquièrent fréquemment la capacité de surmonter et exploiter les barrières chromatiniennes pour contourner leur destin, devenir prolifératives et acquérir des identités aberrantes. La régénération est donc une épée à double tranchant, assurant à la fois l’intégrité des tissus mais étant aussi une voie potentiellement ouverte pour la transformation cancéreuse. Notre objectif est de comprendre les bases épigénétiques de la plasticité cellulaire dans la régénération et le cancer, dans le but d’identifier des mécanismes actionnables pour faciliter la régénération tout en empêchant la tumorigénèse. Notre laboratoire étudie ces mécanismes par des méthodes à haute résolution en épigénomique et en microscopie par l’étude de deux cas modèles: la résistance aux traitements dans les cancers et la régénération des pattes chez le grillon.
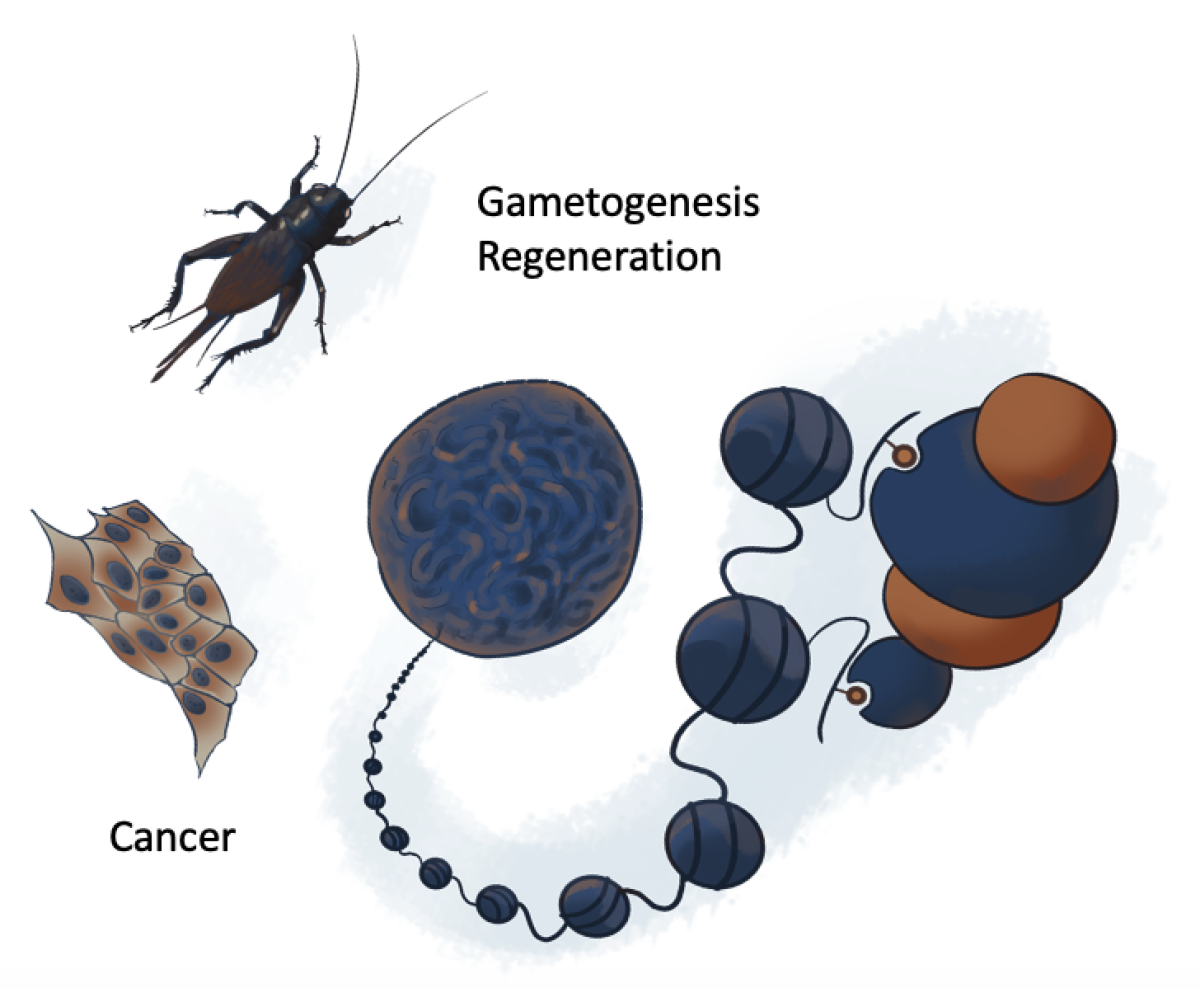
Nos axes de recherche
Les cellules cancéreuses modifient leur épigénome pour échapper au contrôle somatique. Avec un focus sur le cancer du sein, nous voulons comprendre comment cette adaptation conditionne leur identité et détermine leur agressivité ainsi que leur réponse aux traitements.
Comme nombre d’autres insectes et arthropodes, les grillons ont la capacité de régénérer entièrement leurs pattes suite à une amputation. Nous voulons comprendre les mécanismes épigénomiques permettant aux cellules de changer d’identité plusieurs fois pour assurer la bonne tenue de ce processus spectaculaire.
Pendant la différentiation des spermatozoïdes, la chromatine subit des bouleversements majeurs conduisant à l'ultra-compaction du noyau. Ces processus reposent sur une dynamique orchestrée de modification, remplacement et enlèvement des histones et leur remplacement par des protéines spécifiques su spermatozoïde, dont notamment les protamines. Malgré leur importance, ces mécanismes restent une boite noire majeure dans la biologie développemental que nous nous efforçons d'éclairer en combinant des approches génétiques, biochimiques, en microscopie en haute résolution et de modélisation biophysique.
Nos publications majeures
Voir toutes les publicationsNos activités en images
Nos technologies
- Cut&Run
- MNase-seq
- Nanoscopie STORM
- Bioinformatique
- Modèles insectes