Nos axes de recherche
Biologie Structurale de Nouvelles Cibles dans les Maladies Humaines
Dynamique structurelle de protéines et d’ARNs
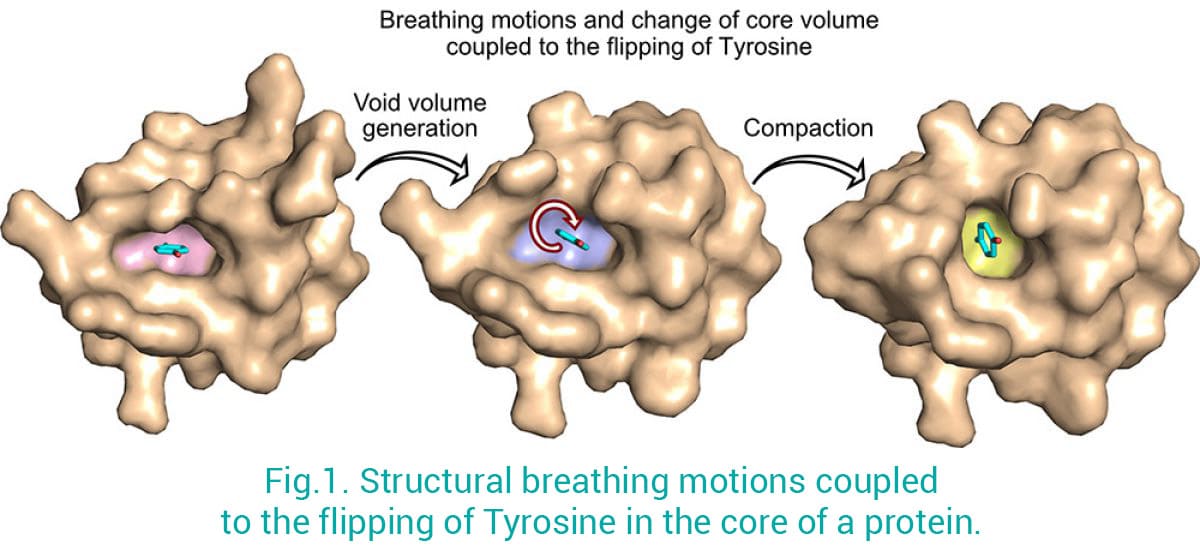
Les protéines ne sont pas des structures rigides : elles bougent et s'adaptent à leur environnement afin d'optimiser leurs formes et leurs fonctions. Nous combinons la cristallographie aux rayons X/Cryo-EM avec la spectroscopie RMN en solution (collaboration avec Jensen’s Lab) pour fournir une image complète des propriétés dynamiques structurelles des macromolécules biologiques. Cette approche s'est avérée extrêmement puissante pour mieux saisir les propriétés ultra-dynamiques des macromolécules en solution qui sont autrement difficiles à aborder en utilisant uniquement les techniques de cristallographie ou de microscopie électronique. Par exemple, en utilisant cette stratégie, nous avons pu résoudre une question de longue date en biologie : comment les résidus aromatiques dans le cœur des protéines peuvent-ils tourner complètement (180 degrés) tout en étant engagés dans des interactions cruciales qui maintiennent le repliement des protéines et assurent ainsi leur fonction ? Nous avons montré les réarrangements structurels, connus sous le nom de "mouvements de respiration", qui sont nécessaires pour le retournement d'un résidu aromatique dans le cœur d'une protéine repliée (Fig. 1) (Mariño Pérez et al 2022). Cela a des implications à la fois pour la conception des protéines et la prédiction de la structure en soulignant comment de petits changements dans le cœur de la protéine peuvent conduire à des changements structurels majeurs, et apporte de nouvelles perspectives sur la façon dont ces petites altérations peuvent conduire à des maladies.
AminoAcyl-ARNt synthétases (AARS)
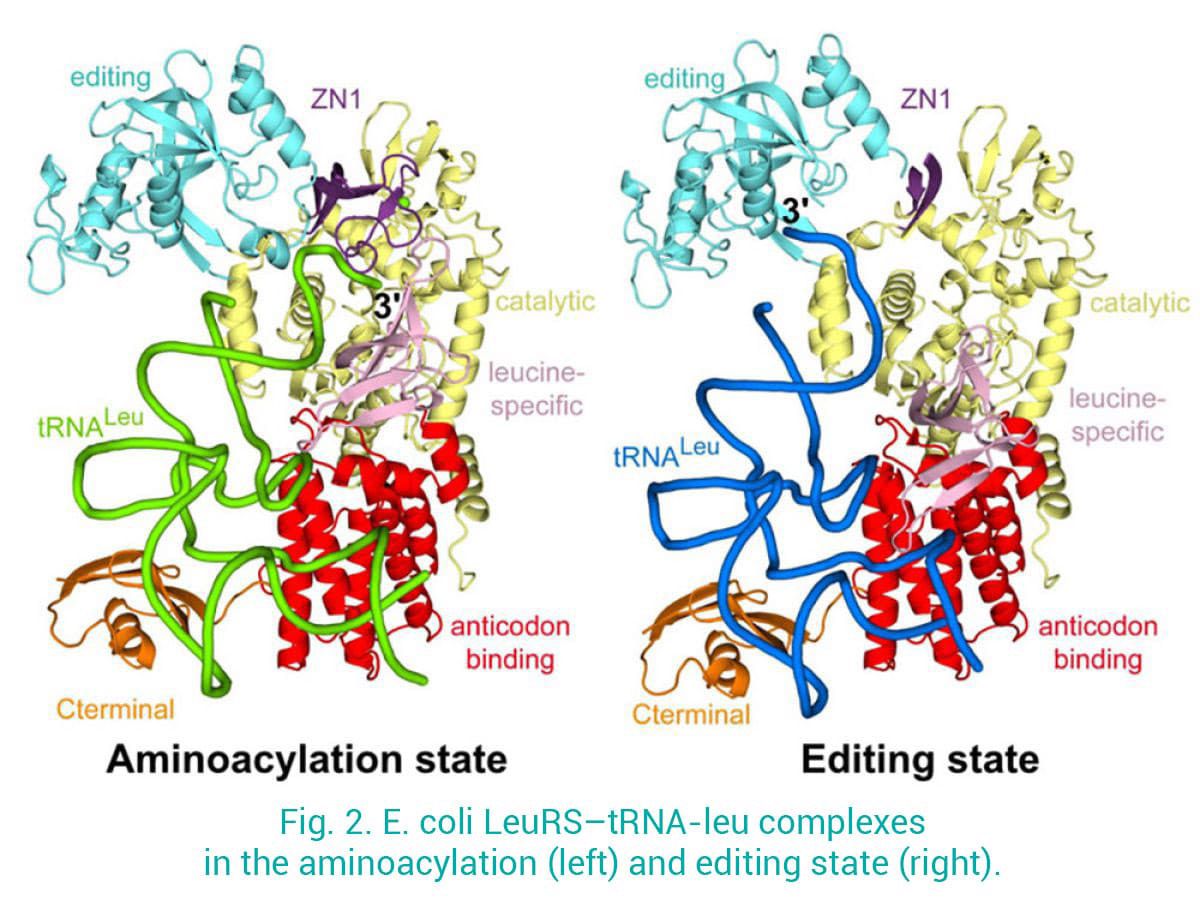
Nous nous intéressons notamment aux Leucyl-tRNA synthétases (LeuRS), qui chargent la leucine sur le tRNA-Leu correspondant et empêchent l'ARNt de se décharger sur des acides aminés chimiquement proches. Nous avons déterminé les structures de LeuRS dans différents états fonctionnels : 1) l'état d'aminoacylation, où l'ARNt 3' est dans le site de synthèse prêt à accepter l'adénylate leu-AMP ; 2) l'état d'édition post-transfert, où l'ARNt 3' mal chargé est lié au site d'édition pour la correction. Cela a permis d'obtenir des informations sans précédent sur la dynamique structurelle et la translocation de l'ARNt 3' entre le site synthétique et le site d'édition, séparés de plus de 30 Å (Palencia et al. 2012, Chopra et al. 2013) (Fig. 2).
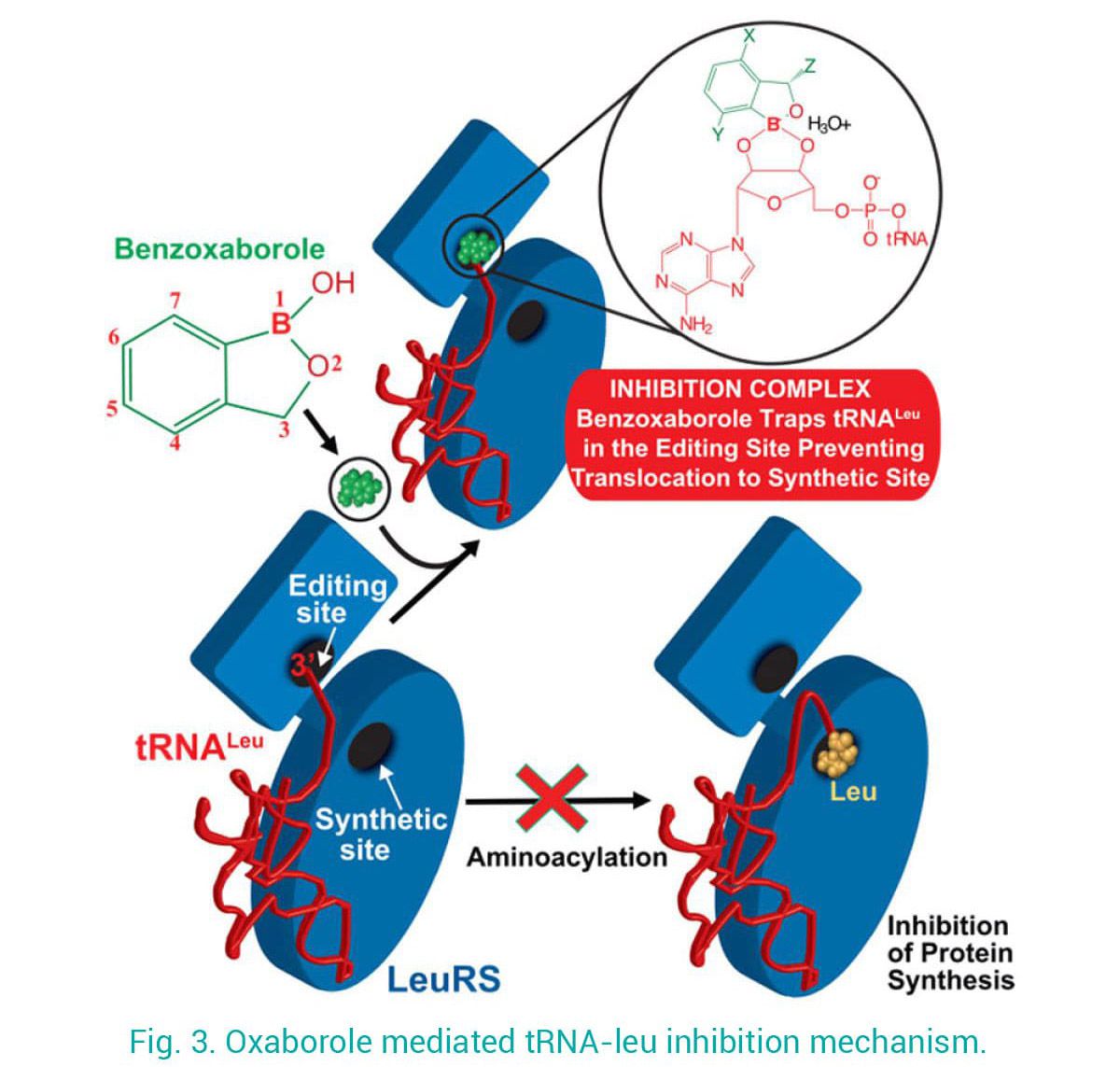
En utilisant ces connaissances mécanistiques, en étroite collaboration avec des équipes universitaires et industrielles, nous avons contribué au développement d'antimicrobiens ciblant LeuRS. Nos structures de LeuRS-ARNt lié à des benzoxaboroles ont dévoilé le mécanisme d'inhibition des nouveaux antibactériens, par lequel l'ARNt est piégé au niveau du site d'édition et inhibe efficacement la synthèse des protéines (Fig. 3). En utilisant une approche de conception de médicaments basée sur la structure, nous avons fait passer la puissance des premiers résultats de micro à nano-molaire et amélioré la sélectivité, ce qui a permis leur progression vers les essais cliniques. En exploitant ce mécanisme d'action, de nouveaux anti-infectieux contre les parasites apicomplexes ont été développés.
Mécanismes macromoléculaires impliqués dans la maturation de l'extrémité 3' des ARNm et la polyadénylation
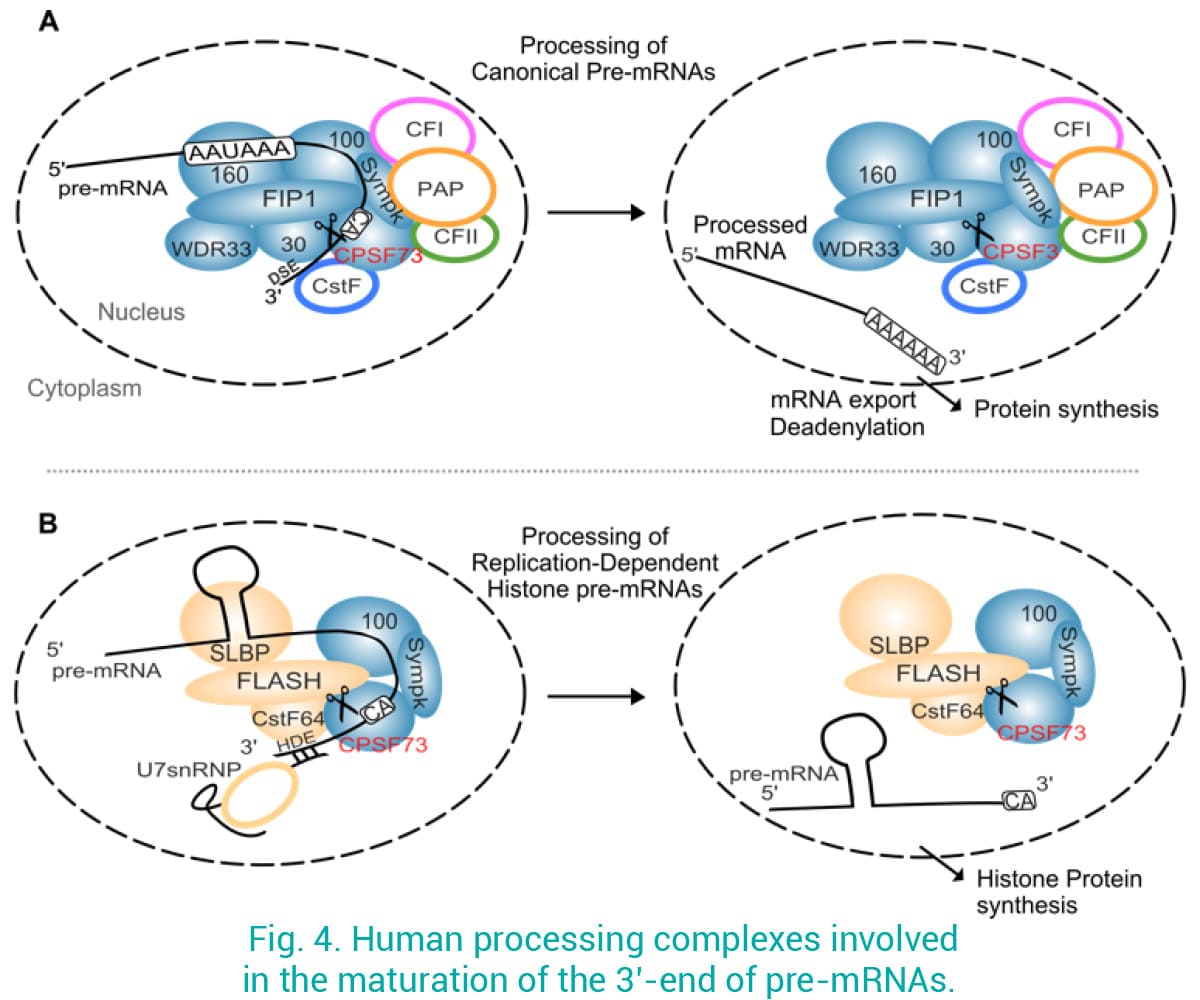
Malgré les preuves croissantes montrant que les défauts dans la maturation de l'ARNm 3' conduisent à des maladies, les facteurs de maturation de l'ARNm sont peu explorés en tant que cibles thérapeutiques. Nous avons récemment montré que le ciblage de CPSF3 par les oxaboroles dans plusieurs pathogènes représente une excellente approche pour traiter les infections (Palencia et al 2017 ; Swale et al 2019). La protéine homologue chez l'homme, appelée CPSF73, s'associe en un grand complexe pour cliver les pré-ARNm 3' canoniques avant l'ajout de la queue poly-A par une polyadénosine polymérase (PAP) (Fig. 4A). CPSF73 est également recruté dans un complexe différent impliqué dans la maturation des pré-mRNAs d'histone dépendants de la réplication (Fig. 4B) (Ielasi et al 2022), qui ne sont pas polyadénylés, un processus étroitement régulé au cours du cycle cellulaire. De plus, nous avons la preuve que le CPSF73 humain est fortement régulé dans de nombreux cancers, et que sa surexpression est corrélée à un mauvais pronostic pour les patients. Nous utilisons une approche interdisciplinaire en combinant la bioinformatique, la biologie des cellules cancéreuses, la biologie structurale et les techniques biophysiques pour étudier les mécanismes associant CPSF73 au cancer, et l'inhibition de CPSF73 par des composés comme nouvelle approche thérapeutique.
Complexes eucaryotes impliqués dans la maturation des pré-ARNt en 3'
Afin de remplir leurs fonctions, les précurseurs d’ARN de transfert (pré-ARNt) doivent être transformés en ARNt qui contiennent une séquence et une longueur spécifiques des extrémités 3' et 5'. Ce mécanisme de transformation des ARNt consiste en 1) un clivage de l'extrémité 5', 2) un clivage de l'extrémité 3', 3) l'ajout d'un bras accepteur CCA à l'extrémité 3', et 4) des modifications post-traitement (effectuées dans le cytoplasme).
Pour les quelques pré-ARNt contenant des introns, deux complexes supplémentaires ont lieu pour scinder l'intron et fusionner le reste. Les étapes 1) et 2) se produisent à la fois dans le noyau ou dans la mitochondrie selon l'isoacceptor d'ARNt.
Nous étudions la maturation nucléaire des pré-ARNt et visons à compléter les données structurelles limitées sur ces processus, ce qui sera utile pour le développement d'applications thérapeutiques.