Équipe
Epissage des ARN, Signalisation cellulaire et Réponse aux thérapies
Dpt: Microenvironnement, Plasticité cellulaire et Signalisation
Nos activités de recherche
Béatrice EYMIN
Chef d’équipe
04 76 54 94 76
Nos axes de recherche
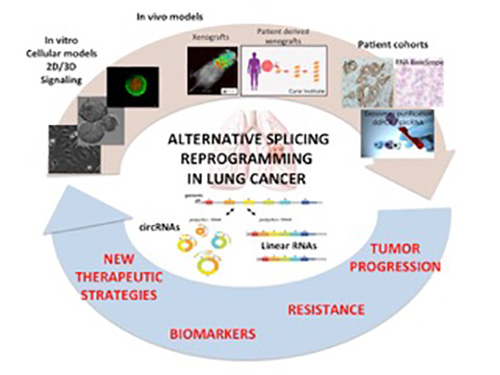
Les protéines SR sont des facteurs d’épissage cruciaux dont nous avons décrit précédemment la surexpression dans les tumeurs du poumon. Nos objectifs sont d’approfondir le rôle de ces protéines dans la progression des tumeurs pulmonaires et leur échappement thérapeutique. Plus spécifiquement, nous voulons déterminer si et comment ces protéines contribuent à l’instabilité génomique et aux mutations au cours de la résilience thérapeutique des cellules cancéreuses pulmonaires en régulant la réponse aux dommages de l’ADN et la réparation de l’ADN.
En savoir plusLes inhibiteurs pharmacologiques ciblant différents composants du splicéosome constituent un domaine thérapeutique prometteur dans le cancer. Cependant, les mécanismes moléculaires soutenant leurs effets anticancéreux restent largement méconnus. Nos objectifs sont de déterminer si l'utilisation de ces inhibiteurs, seuls ou en combinaison avec des chimiothérapies, pourrait être un moyen de contrer la résistance aux médicaments dans le cancer du poumon.
En savoir plusLe récepteur EGFR joue un rôle clé dans la progression du cancer du poumon, et les inhibiteurs de son activité tyrosine kinase (EGFR-TKI) constituent une approche thérapeutique très prometteuse avec des résultats sans précédent sur la survie des patients. Cependant malgré une efficacité thérapeutique prouvée, des résistances se développent inévitablement. Nos objectifs sont d'évaluer la contribution de la reprogrammation de l'épissage de l'ARN au développement de la résistance acquise aux EGFR-TKI.
En savoir plusLes ARNs circulaires (ARNcirc) sont une nouvelle catégorie d’ARNs majoritairement non codants, très stables et détectables dans les biopsies liquides, en faisant des biomarqueurs prometteurs. Ces dernières années, leur rôle dans l’initiation et la progression des cancers ainsi que dans la réponse aux traitements a émergé, mais les mécanismes moléculaires impliqués restent largement méconnus. Nos objectifs sont d’identifier des ARNcirc contribuant à la résilience thérapeutique dans les cancers du poumon.
En savoir plusNos publications majeures
Voir toutes les publicationsNos activités en images
Nos collaborations
- Pr Ester Hamond (Institute for Radiation Oncology, Oxford, United Kingdom)
- Dr Pablo Huertas (Andalusian Molecular Biology and Regenerative Medicine Centre, CABIMER, Seville, Spain)
- Dr Eleni Nikolakaki (Laboratory of Biochemistry, Aristotelian University, Thessaloniki, Greece)
- Dr Didier Auboeuf (ENS, Lyon)
- Dr Didier Decaudin (Institut Curie, Paris)
- Dr Martin Dutertre (Institut Curie, Paris)
- Dr Carmen Garrido (U1231, Dijon)
- Pr Nicolas Girard (Institut Curie/Montsouris, Paris)
- Dr Antonio Maraver (IRCM, Montpellier)
- Dr Franck Mortreux (ENS, Lyon)
- Dr Olivier Sordet (CRCT, Toulouse)
Nos technologies
- 2D and 3D cell culture
- Proximity Ligation assay (PLA)
- RNA BaseScope
- RNA sequencing
- RT/PCR and quantitative PCR (QPCR)
- JESS
- Western blotting
- DNA Fiber assay
- Immunoprecipitation
- GST-pull down
- Immunofluorescence and Immunohistochemistry
- Flow cytometry
- Seahorse
- SIRF